🚅 大概今年的3-4月份,我尝试自己从头预训练了一个单细胞转录组大模型STELLA,有人类和小鼠两个版本。由于市面上现有单细胞转录组大模型数量太多(Geneformer,scGPT,scFoundation,LangCell,Cell2Sentence等),这个模型预感可能发表不出去了😢。这个项目就当做自己的一个练手项目吧……🥲🥲🥲。整个训练流程完全基于huggingface生态进行开发,数据集构建采用datasets模块,模型训练使用transformers的Trainer,自己也是第一次手搓大模型,也是边学习边写,有些过程还是比较难写的(例如DataCollator等),自己也debug了很多代码,学习了很多huggingface代码工程化思想,收益很多。虽然模型可能发表不出去了,但是整个仓库代码个人认为写的还是比较清晰的(某中科院大佬点赞称比已经发表的大模型代码好读很多~😂(bushi)),适合刚入门大模型的同学学习提升自己的代码能力,熟悉整个构建流程。
- 含泪开源,重要的事情说三遍:https://github.com/Peg-Wu/scRNA-LLM
- 含泪开源,重要的事情说三遍:https://github.com/Peg-Wu/scRNA-LLM
- 含泪开源,重要的事情说三遍:https://github.com/Peg-Wu/scRNA-LLM
- 如果觉得项目对你有帮助,麻烦给小弟一个star吧~
大体介绍一下STELLA:
1. 数据
- 预训练数据总量
| scRNA | 人类 | 小鼠 |
|---|---|---|
| 细胞数目 | 109,700,701 | 84,057,580 |
| 基因词库大小 | 42570 | 36343 |
- 预处理:normalize_total (1e4) + log1p,接着使用TDigest算法对基因表达值进行全局分箱(100 bins)
2. 模型
- 模型输入:基因symbol序列 + 分箱后的基因expression序列(embedding相加)
- 模型架构:相当于双向Llama,把FFN层换成了DeepSeekMoE混合专家结构
- 预训练任务:对分箱后的基因表达值进行掩码预测
4. 预训练效果
- 细胞类型注释 & 基因调控网络推断(GRN)
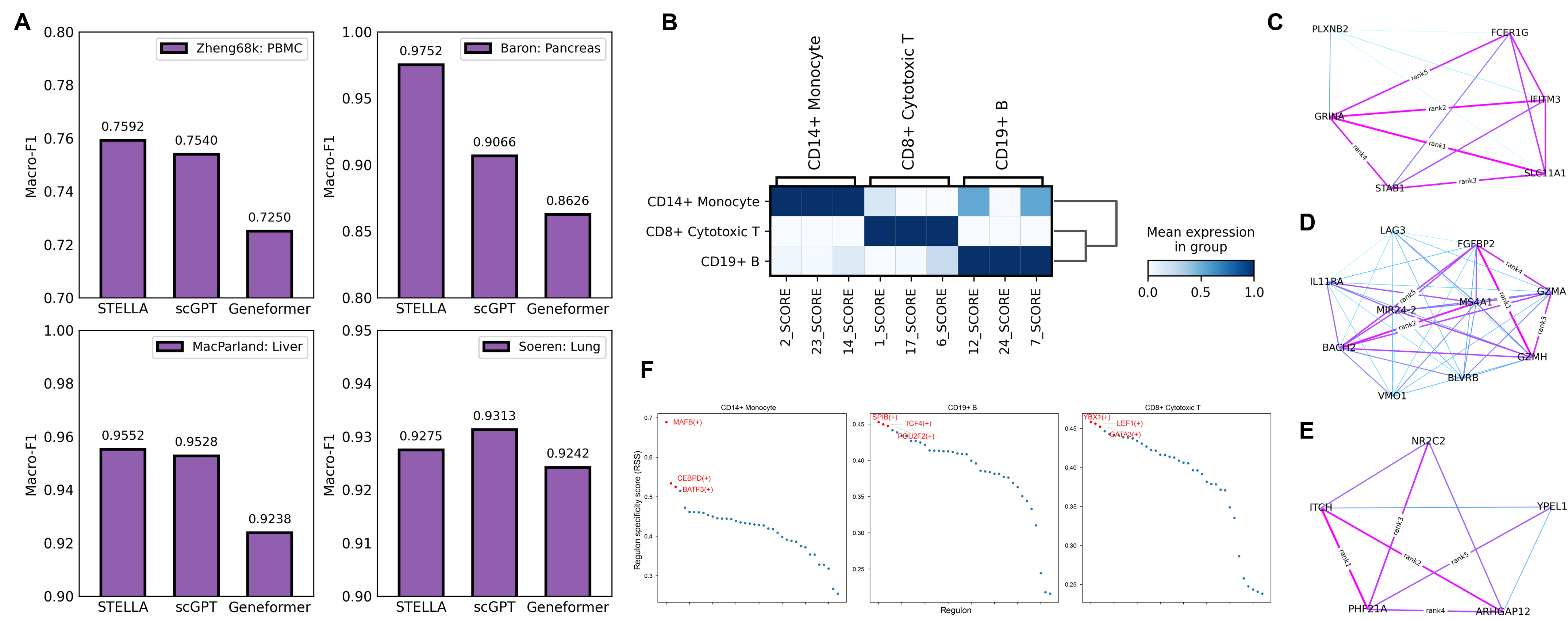
- 总体效果和scGPT差不多,细胞类型注释任务上胰腺数据集STELLA表现要更好
- GRN就是一些大模型常做的任务,利用Gene Embedding找细胞类型特异性的Gene Set;接PySCENIC预测细胞类型特异性的转录因子;基于Gene Embedding的余弦相似度构建基因共表达网络等
- 单细胞基因扰动预测
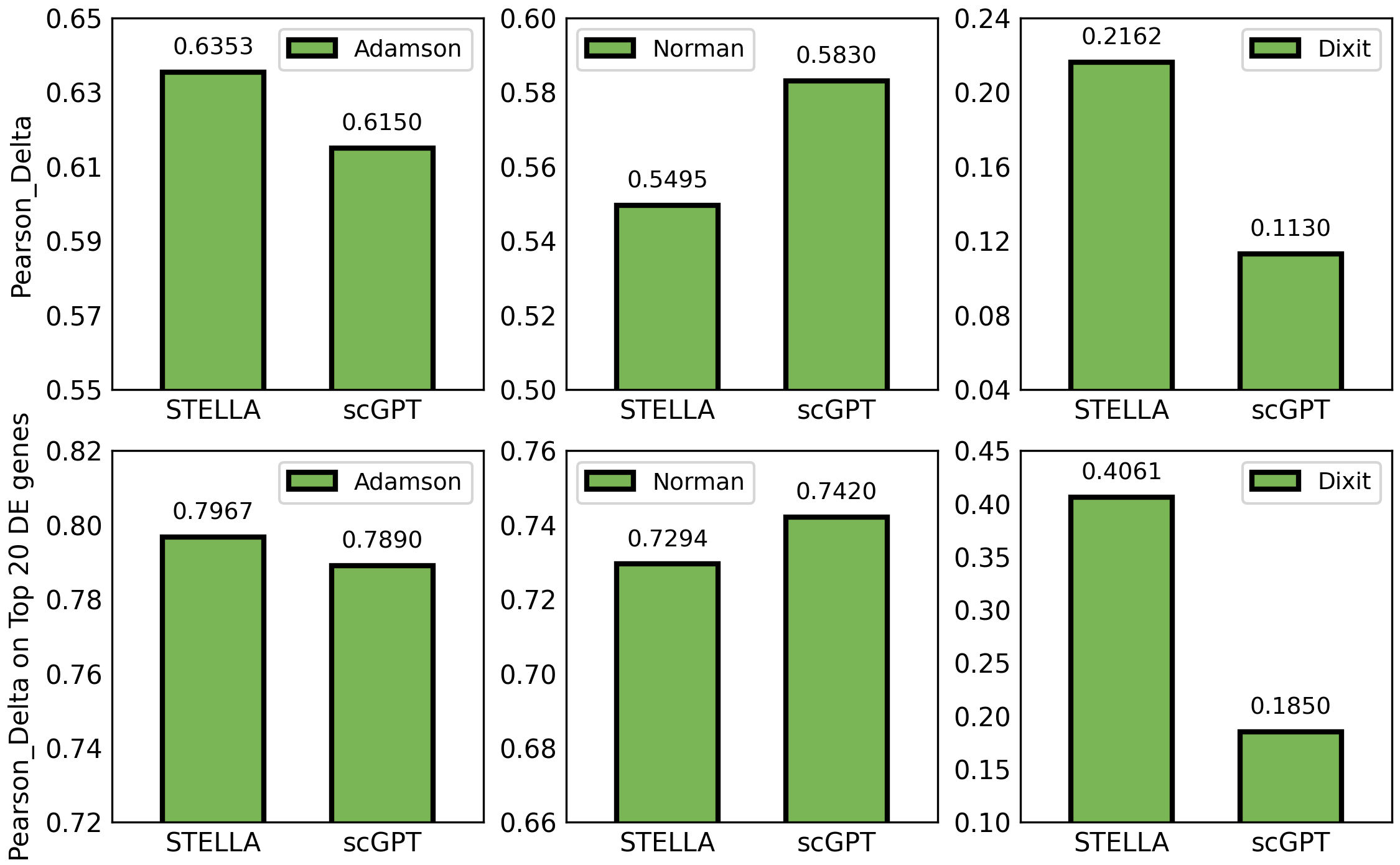
- 熟悉基因扰动预测这个领域的都知道,单细胞大模型在这个任务上的表现纯一坨💩,预训练参数没有一点作用
- 我个人觉得这个任务表现差纯纯是数据集的问题(几乎全是噪声),和模型一点关系都没有,感觉这个领域(现在也被叫做虚拟细胞),就是领域内的人自嗨hhh